
| 目标序列捕获技术 |
|
1. 前 言 人类基因组计划(Human Genome Project, HGP)给基因组学研究带来了天翻地覆的变化,通过测定人类基因组DNA的序列,探寻基因在染色体上的位置,明确基因的结构和功能,解读人类的全部遗传信息,人类第一次在分子水平上全面认识自我。在这期间,建立了两项非常重要的高通量技术:基因芯片和新一代测序技术。这两种技术进一步结合,就产生了一种新的解决方案:目标序列捕获测序技术。新一代测序技术的发展和成功应用使得普通实验室对整个人类基因组进行测序成为可能,由此,科学家们提出了千元人类基因组计划。然而就目前而言,全基因组测序对实验成本和能力的要求还是很高,并且由于大部分基因的功能还不是很清楚,对所产生的海量数据进行生物信息学分析将是一项巨大的挑战。 在新一代测序技术以前,对基因组大片段特定区域的研究主要通过PCR扩增后进行传统毛细管测序的方法。这种方法的问题是目标片段大于500kb时PCR和测序的成本会变得让研究人员难以承受。而传统的单基因疾病的解决方案是家系连锁定位,最后定位的区域往往是以兆(M)bp来计算的,这就为后续的研究工作带来了困难。 近年,目标序列捕获、全外显子捕获等方法能更加经济有效的靶定基因组的相应区域。而且这些方法产生的大量DNA片段非常适合使用新一代测序平台进行测序。通过这些技术的组合,人们对于遗传疾病的研究的效率大大的提高,2009年-2011年,应用该技术解决遗传疾病的文章就达到200余篇。 基因捕获测序的主要优势在于可针对特定区域进行测序,有效降低了测序成本,提高了测序深度,更精确地发现特定区域遗传变异信息。目前常用的基因捕获方法包括杂交捕获和多重PCR扩增,核心技术主要掌握在国外大生物科技公司手上,如Roche、Agilent、ADV、IDT、Life Technologies等。其中,杂交捕获的特点是能够对外显子组甚至更大的目标区域进行捕获,但操作流程复杂,需要依赖较多专门的仪器设备;多重PCR则操作简单灵活,对仪器要求最小化,能在数小时内完成目标序列富集和文库构建,适用于相对较小的目标序列捕获。 2 目标序列捕获测序技术 2.1 目标序列捕获 目标序列捕获是指通过某种方法有选择性的分离或者富集基因组的特定片段。从这个定义看,目标片段的PCR也是目标序列捕获的一种,不过这种方法通量小,目前一次PCR获取最长的基因组DNA片段长度应该不会超过50kb,而且需要特殊的酶和特殊的PCR条件,成本高昂,稳定性差。当然100kb以下的片段,通过多次PCR的方式也是可行的,一般每个扩增片段的长度在500bp左右性价比最佳。 目标序列捕获的另一种重要的方法是根据核酸分子碱基互补杂交原理发展的。即根据目标基因组序列,设计与之完全互补的探针,将这些探针固定在某些支持物上(用于分离),然后打断基因组DNA,加上接头(用于测序)后与探针杂交,洗脱未杂交上的DNA,回收目标DNA片段,可以直接建库进行DNA测序。根据杂交时状态不同,目标序列捕获可以分为固相杂交法和液相杂交法。固相杂交法所用的探针通常都是固定在固体支持物上,如玻璃、塑料等,其中最典型的是基因芯片。基因芯片的两大厂商(Agilent,NimbleGen)都提供这一方法的商业化试剂盒。固相捕获系统构建如下,首先选定目标DNA区域,在修饰了的玻璃片基上原位合成一系列与目标区域互补的探针。通常,基因芯片是杂交后,洗脱未杂交上的DNA片段,然后扫描成像获取每个探针的杂交信号,而固相杂交捕获则只有最后一步不同,即与探针杂交的DNA被洗脱下来,用于后序的测序工作。 液相杂交与固相杂交最大的差异在于杂交反应的环境不同。液相杂交是通过在溶液中,目标DNA片段和已带有生物素标记探针直接杂交,然后通过生物素亲和素的反应使目标DNA片段锚定在带有亲和素的微珠上。洗去非目标DNA,洗脱后,富集的DNA用于测序。液相杂交与固相杂交相比有两大优势:第一、杂交效率更高;第二、易于操作,时间短,便于自动化操作。使用这种方式的典型产品是安捷伦(Agilent)公司推出的SureSelect目标序列捕获系统(图1)。 图1 安捷伦SureSelect 目标序列捕获系统流程示意图(引用自agilent网站)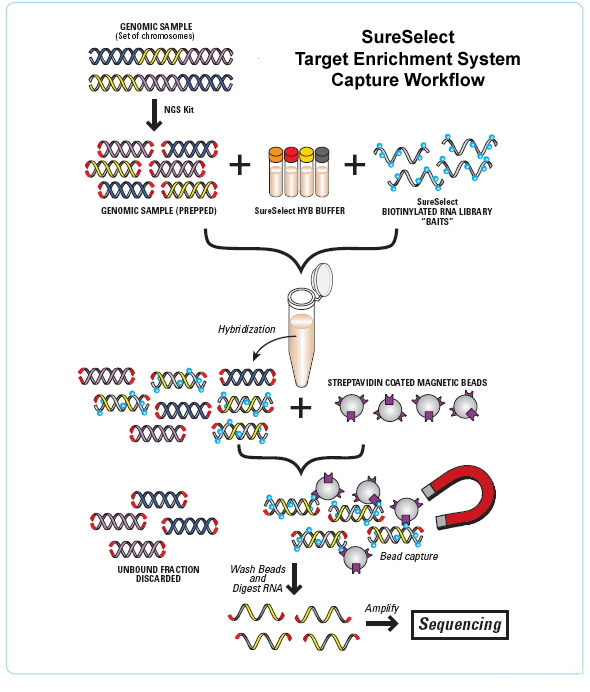 构建一个基于杂交原理的目标序列捕获系统,有两点需要考虑,即探针的长度和探针的合成成本。一般来说一个8碱基的探针就有了足够的杂交特异性,而探针越长,杂交的特异性就越差。目前商业试剂盒的探针长度都在60nt到200nt之间,这其中的一个重要考虑是,杂交的特异性限定(或者说杂交的错配容忍度)。我们需要研究的是目标DNA片段中发生的突变、插入/缺失等,如果探针特异性太高,在DNA捕获时就会产生有利于参考序列(与探针完全互补的序列)的选择,这在后期数据统计上就会产生明显的偏差,而探针太长又会有太多的非特异杂交,非目标序列会急剧增加。 长探针合成的成本也是制约这种技术发展的关键因素之一,比如说设计1万条100nt长带有锚定物(生物素)的引物探针,传统的引物合成需要非常高昂的成本。对于基因芯片厂商来说,这是非常容易的事情,这得益于他们的探针原位合成技术。原位合成探针的低成本和灵活性非常有利于特定区域的基因组捕获,Agilent公司提供了客户定制的系统,DNA捕获区域从200kb到5.8Mb都可以。 2.2全外显子组捕获我们知道编码蛋白的外显子仅占人类基因组的1%左右,但是它非常重要。目前外显子组捕获技术已发展成熟。这些技术都是基于杂交的原理,包括基于阵列的固相杂交和液相杂交。目前安捷伦公司发展的全外显子捕获试剂盒(SureSelect)最高捕获量为50Mb。罗氏公司(Roche NimbleGen)同时推出了基于固态杂交法(2.1M SeqCap array)和液相杂交法( SeqCap EZ)两种技术。 当然,“人类全外显子组”有很多种不同的定义,两家公司提供外显子组捕获的商品化试剂盒,除了靶向到大约30M编码蛋白的外显子区域外,两家公司也提供靶向选择miRNA编码区的试剂盒,并且可以加入自主选择的捕获区域(安捷伦公司)。这些产品捕获的DNA序列虽然只是全基因组的部分区域,但在成本和时间花费上具有很大的优势。 2.3 染色体区域捕获人的基因组并不是从头到尾一条DNA链的结构,实际上是由22对常染色体和1对性染色体构成,这使我们单独分离某条染色体后测序成为了可能。由于每条染色体都有自己特定的形态和结构,所以可以通过专业的设备识别每条染色体,分离后染色体构建成DNA文库后就可以用新一代的测序技术进行深度测序了。这一策略非常有效,已有中期染色体经显微切割捕获后测序的案例被报导。虽然这些方法都需要高度专业的仪器设备,但是对于某些独特的生物学问题确实是非常有用的。 3. 目标序列捕获技术的应用 3.1 在医学研究中的应用目标序列捕获实验的主要用途之一是研究导致疾病发生的遗传变异。对于单基因遗传疾病,传统的正向遗传手段是通过家系连锁分析。它要求有足够的分子标记,当然自从SNP芯片出现后此问题得到极大的缓解。但是传统方法还有一个缺点是要求研究的家系有足够多的交换(至少三代),但是由于患病家系个体的寿命短或者子一代到一定年龄后才发病,导致大量家系只能收集到两代人的样本。而用外显子组捕获测序来研究这些案例就可以解决这个问题,因为它只需要核心家系就可以了。而且理论上全外显子组捕获实验对于多基因疾病同样有效。最近已经有多篇报导利用捕获加测序的方法研究遗传疾病,并取得了不错的结果。 Ng 等利用基于微阵列的杂交技术对12个人的外显子组进行了测序。其中4个个体是无亲缘关系的弗里曼-谢尔登综合症患者,另外8个个体是正常对照,来源于国际人类单体型图计划(HapMap)。弗里曼-谢尔登综合症已知是由MYH3基因的变异引起的孟德尔显性遗传疾病,非常罕见。这一研究结果发表在2009年的Nature杂志,是第一篇人类外显子组测序的论文。该文章的发表为单基因遗传病研究提供了全新的方法,同时也建立了完整的测序数据分析流程:a.去掉同义突变;b.去掉公共数据库正常人携带的SNP位点;c.通过软件预测突变对所表达蛋白功能的影响;d.找出患者共同拥有的突变,最终定位到了候选致病基因MYH3。 Ng 等在Nature Genetics在线发表了第一篇利用外显子捕获测序技术寻找到未知病因的致病基因的文章。研究人员选择了三个独立的米勒综合症家系,对其中4名患者进行外显子测序。通过与人类参考序列比较,4个患者的DHODH基因都产生了变异。通过进一步验证,研究人员在其它3个家系的米勒综合症患者中发现DHODH基因上存在同样突变。这篇文章为研究未知病因的单基因遗传病建立了外显子捕获测序技术的解决方案。 在肿瘤疾病的研究方面,采用全外显子捕获测序技术也取得了重要的成果。葡萄膜恶性黑色素瘤(maligment melanoma of uvea)是一种较多见的恶性眼内肿瘤,Anne Bowcock等采用了外显子组测序方法,结果发现在31个肿瘤样本中有26个(占84%)样本的BAP1基因存在着失活性突变。Simon等将此技术应用在胰腺神经内分泌瘤(PanNET)的突变检测上,发现在68位被检测的患者样本中, MEN1基因突变有30例,DAXX突变有17例,ATRX突变12例。瑞金医院陈竺院士/陈赛娟院士的研究小组利用外显子组测序技术对急性髓系白血病M5型的患者血液样品进行了筛查,发现112名患者中有23例存在DNMT3A突变,比例为20.5%。 全外显子组捕获测序还是传统家系定位的有益补充,两者结合将更快更有效锁定致病位点。安徽医科大学张学军教授研究逆向性痤疮(Inversa acne, AI)就是两种研究方法结合的典型案例。该团队刚开始采用全基因组扫描结合连锁分析的方法将致病基因定位于1p21.1-1q25.3区域,由于该连锁区域包含大约900个基因,范围较大,后续很难进行进一步的精细定位。他们采用了外显子测序技术解决了这一难题。通过外显子捕获测序结合连锁定位的结果,证实NCSTN基因与逆向性痤疮的发生相关。 全外显子捕获技术的出现,将极大的推动人们对基因和疾病关系的研究。根据统计,大概85%的单基因疾病突变位点位于外显子区域,与全基因组重测序相比,相同成本下,覆盖度更深、数据准确性更高,更加简便、经济、高效。它也可用于寻找复杂疾病如癌症、糖尿病、肥胖症的致病基因和易感基因等的研究。同时,外显子的变化和其表达的蛋白直接相关,科学家们能够结合现有资源非常直接的解释研究结果。 3.2 其他研究应用目标序列捕获技术在生物进化的研究中也起着重要的作用。例如,在研究古代尼安德特人的基因组中。由于尼安德特人已经灭绝已经有几万年,遗留下来的只有骨骼,DNA已经严重降解。这些DNA和新鲜样本的DNA比,处理非常困难,同时还存在着非常严重的微生物基因组污染,因此这种样品是不适合用鸟枪法测序的。研究人员运用目标序列捕获技术对尼安德特人基因组中有关人类进化相关的基因进行富集,富集率高达190,000倍,这样就获得了尼安德特人序列中几乎所有感兴趣的位点。后来研究人员确定了88个在人类中已经被修复的替换,这些发现可以告诉我们人类和尼安德特人在分子水平有何不同。此项研究过程中建立的方法学可应用于考古学、古生物学乃至人体法医学等领域中,比如恐龙基因组的研究等。 外显子组捕获也已经被用于研究近代的进化变异。研究人员应用全外显子测序技术,对青藏高原世居藏族人群和低海拔人群进行比较,发现了藏族人群适应高原环境的关键基因EPAS1。通过对藏族人群中EPAS1基因的改变位点进行关联分析,发现EPAS1基因中受选择的基因型与藏族人群血红蛋白的代谢有关,藏族人群EPAS1基因阻止了血红蛋白浓度在高海拔区域过度升高,降低了各种高原性疾病发生的可能性。同时根据数据分析发现,汉族(北方)与藏族的基因非常相近,其最大的区别是EPAS1基因的不同,而这作为汉藏分离的证据可以测定出分离的年代(汉藏分离大概在2750年左右)。这篇文献表明外显子组捕获测序技术在等位基因频率的研究中是准确、有用的,尤其对于那些SNP芯片中未包含的罕见SNP的研究更为有用。通过外显子组捕获技术研究近代和古代的遗传差异,我们能够更加全面的了解我们的进化历史。 目标序列捕获测序技术也可以用来研究基因组的拷贝数变化。拷贝数变化是遗传疾病的另一个重要原因。目前拷贝数变化的研究工具主要是基因芯片,如arrayCGH芯片或者SNP芯片等。这些方法通过杂交信号强度来区分拷贝数,属于“模拟”信号,其分辨率取决于芯片上探针的密度。Conrad等利用目标序列捕获测序这一高分辨率技术研究了特定拷贝数变异区域,检测到了真正的“断点”,并推测有些断点可能于修复机制有关。作者指出这种方法用于研究简单区域的CNV是非常有用的,但是对于那些有很多重复的复杂的基因组运用此技术,无论是捕获探针的设计还是测序的分析都具有挑战性。 随着技术的发展,目前捕获技术不仅限于基因组DNA,已经有研究利用捕获测序技术研究RNA序列。Levin等利用此技术富集感兴趣基因的RNA,通过对这些基因的有效的富集,研究人员不需要增加测序总量就可以检测低丰度基因,同时他们还检测到了基因融合。利用RNA捕获测序技术可研究许多问题,如低丰度转录本的定量,基因的可变剪切,基因融合以及等位基因的表达。安捷伦公司于今年初推出了SureSelect RNA捕获试剂盒,相信通过该试剂盒结合新一代测序技术将极大的推动转录组的研究。 4. 展望目标序列捕获测序是目前基因组学研究中的一个热点技术,主要原因是全基因组测序需要耗费大量的成本和时间。所以有选择性(目标序列捕获)的深度测序是目前基因组研究的明智选择,当然不断改进的测序技术和不断改进的生物信息分析将会大幅度的降低成本和时间。当人们只对部分基因组进行测序时,在相同成本下,研究者可以研究到更多的样本数量和测到更深的深度。我们知道,样本数量是发现致病基因的关键指标,尤其是较常见的疾病,样本量越多,定位到疾病相关基因的可能性越大。特别对于一些稀有的变异或者部分体细胞的基因突变,测序深度决定了靶向测序是一种有效的工具。 当全基因组测序的试剂成本真的降到1000美元时,处理随之而来的海量生物信息将是巨大的问题,分析的费用也会远远高于1000美元。如果同时解释个体的所有遗传信息(DNA序列,突变,CNV,甲基化,转录本等),研究人员能否从获取的数百万位点中挑选出重要的突变位点?重复的序列如何屏蔽?各组学间数据如何交叉分析?结果如何解释?这些都需要新理论和新算法的出现,然而对于疾病的遗传研究时不我待。因此,靶向测序作为全基因组测序的补充技术是非常有用的,它大大简化了分析的目标。 分子诊断市场无比广大,但是诊断市场对成本的要求更为苛刻,至少全基因组测序在很长的一段时间内无法进入分子诊断市场,这就是目标序列捕获测序的机会。例如DMD基因,长度2500kb,由79个外显子和78个内含子构成,其突变是引起杜氏进行性肌营养不良病的原因,非常适合采用靶向测序。还有一些遗传病在表型上非常相似,但可能是目前已知基因变异的一种,这样我们也可以把这些变异区域并在一起,设计目标序列探针并捕获测序,这比传统的PCR后毛细管测序通量高,速度快,深度深。 目标序列捕获技术已被证明是一个强大、有效的技术,并在新一代高通量测序中发挥独特之处,已经产生了许多令人兴奋的新发现,应用领域也越来越广泛。 |